Regensburg
In Anlehnung an die Genomik, die sich mit der Analyse der Gesamtheit der von Generation zu Generation vererbbaren Information von Lebewesen beschäftigt, setzen sich Proteomik und Metabolomik die Identifizierung und Quantifizierung aller Eiweißmoleküle und Stoffwechselprodukte in Geweben und Körperflüssigkeiten wie Blut und Urin zum Ziel. Im Gegensatz zum Genom, dessen Zusammensetzung während eines Lebens nahezu unverändert bleibt, ändern sich die qualitative und quantitative Zusammensetzung von Proteom und Metabolom kontinuierlich in Reaktion auf genetische und nichtgenetische Einflüsse. Vor allem die Zusammensetzung des Metaboloms wird nachhaltig durch die im Darm physiologisch vorkommenden Bakterien beeinflusst, die unter anderen für die Niere schädigende Stoffe wie Indole und Kresole bilden. Die große Vielfalt der physikochemikalischen Eigenschaften von Proteinen und Stoffwechselprodukten stellt eine besondere Herausforderung für deren Analyse dar. Daher gibt es im Gegensatz zur Analyse der aus chemischer Sicht einfach gestrickten genetischen Information keine Technik, die in einem Schritt den Nachweis aller Proteine bzw Stoffwechselprodukte erlaubt. Vielmehr kommen eine Vielzahl unterschiedlicher Trenntechniken wie Elektrophorese, Flüssig- und Gaschromatographie, sowie Detektoren, inklusive Kernspinspektroskopie und Massenspektrometrie, zum Einsatz. Die computergestützte Auswertung der Proteom- und Metabolomdaten bildet dann den funktionellen Ist- Zustand eines Lebewesens als Folge genetischer Vorgaben und momentan einwirkender physikalischer, chemischer, biologischer, diätischer und therapeutischer Faktoren ab. Damit ist es nicht nur möglich, die Wirkung von Medikamenten zu beobachten, sondern auch die Wirksamkeit therapeutischer Maßnahmen genauer vorherzusagen als dies derzeit mit konventionellen medizinischen und genetischen Methoden möglich ist.
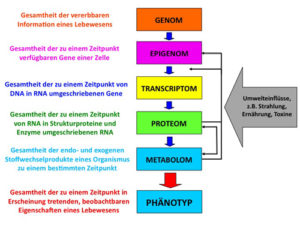
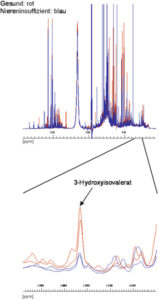
Abbildung von NMR Fingerprints von Urin gesunder (rot) und niereninsuffizienter (blau) Probanden sowie ein Ausschnitt, der den Unterschied in der Konzentration von Stoffwechselprodukten und Peptiden zeigt.